Jak odczytywać tagi w pliku DICOM?
W tym wpisie dowiesz się czym jest standard DICOM i dlaczego tak ważne jest odczytywanie informacji w nim zapisanych. Zapraszam do lektury!
Czym jest standard DICOM?
DICOM to norma opracowana przez ACR i NEMA dla potrzeb ujednolicenia wymiany i interpretacji danych medycznych reprezentujących lub związanych z obrazami diagnostycznymi w medycynie.DICOM znajduje zastosowanie głównie w przetwarzaniu obrazów tomografii komputerowej (TK), obrazowania metodą rezonansu magnetycznego (RM), pozytonowej tomografii emisyjnej (PET), cyfrowej angiografii subtrakcyjnej (DSA), cyfrowej radiografii konwencjonalnej (CR), radiografii cyfrowej (DR) oraz wszystkich wykorzystujących technologie cyfrowe badań o wysokiej rozdzielczości obrazu. Dane w formacie DICOM mają dużą objętość, wymagają specjalnego oprogramowania i sprzętu komputerowego, a także łączy o wysokiej przepustowości, za to pozwalają zachować wysoką jakość obrazu. Stosowanie normy DICOM umożliwia, między innymi, funkcjonowanie teleradiologii.
Tagi w plikach DICOM
Tagi w plikach DICOM (Digital Imaging and Communications in Medicine) są podstawowymi jednostkami danych używanymi do przechowywania informacji w tych plikach. Każdy tag w pliku DICOM składa się z pary wartości: "Grupa, Element", które identyfikują specyficzny element danych. Tagi te są zdefiniowane przez standard DICOM i służą do przechowywania zarówno danych obrazowych, jak i metadanych, takich jak informacje o pacjencie, ustawienia urządzeń, szczegóły dotyczące badania i wiele innych. W tym wpisie dowiesz się jak odczytywać podstawowe tagi DICOM. Odsyłam do pełnej listy tagów https://www.dicomlibrary.com/dicom/dicom-tags/.
Opiszmy sobie kilka z nich.
(0002,0000) File Meta Information Group Length - UL (Unsigned Long) - przechowuje długość informacji o metadanych pliku.
(0002,0001) File Meta Information Version - OB (Other Byte) - zawiera wersję informacji o metadanych pliku.
(0002,0002) Media Storage SOP Class UID - UI (Unique Identifier) - identyfikator klasy SOP (Service Object Pair), która określa rodzaj przechowywanego obiektu.
(0002,0010) Transfer Syntax UID - UI (Unique Identifier) - identyfikator składni przenoszenia, który określa sposób kodowania obrazów i innych danych w pliku.
(0002,0012) Implementation Class UID - UI (Unique Identifier) - unikalny identyfikator implementacji, który identyfikuje oprogramowanie lub system, który wygenerował plik DICOM.
W tym też inne tagi, czyli te, które nas interesują.
Tagi są zorganizowane w grupy, które logicznie grupują podobne dane. Na przykład:
- Grupa 0002: Informacje o pliku
- Grupa 0010: Informacje o pacjencie
- Grupa 0020: Informacje o badaniu i serii
- Grupa 0028: Informacje o obrazowaniu
Każdy tag ma zdefiniowane znaczenie w kontekście danych medycznych. Na przykład:
- (0010, 0010) - Nazwisko pacjenta
- (0010, 0020) - ID pacjenta
- (0008, 0020) - Data badania
- (0008, 0030) - Czas badania
WG-21, czyli grupy robocze w CT
WG-21 opracowuje i utrzymuje obiekty otrzymane w wyniku tomografii komputerowej tak, aby były zgodne ze standardem, w zakresie promieniowania rentgenowskiego, raportów technicznych i innych danych klinicznych. Ta grupa ma możliwość dalszych udoskonaleń w obszarze klinicznym interoperacyjności tomografii komputerowej. Aktualnie jest rozwijanych tylko 18 grup. Osobna grupa będzie do tomografii komputerowej, rezonansu magnetycznego a inna do ultrasonografii. WG z języka angielskiego working group czyli grupy robocze. Więcej na temat tej grupy roboczej odsyłam do linka https://www.dicomstandard.org/activity/wgs/wg-21.
Głownym celem WG-21 jest opracowanie i utrzymanie obiektów specyficznych dla TK dla standardu DICOM w domenach obrazowania nD X-Ray, raportów technicznych, informacji o dawce i informacji klinicznych, które towarzyszą ogólnej procedurze TK pacjenta. Obejmują one (ale nie ograniczają się do) pozyskiwanie, przetwarzanie, przechowywanie, komunikację, wyświetlanie i raportowanie. Grupa CT poszukuje możliwości dalszych ulepszeń w obszarze interoperacyjności klinicznej TK.
Czas na praktykę
Do pełnego odczytu będzie nam potrzebna tylko biblioteka pydicom. Znajdują się tam wszystkie procedury, które są wymagane do poprawnego odczytu. Dokumentacja tej biblioteki znajduje się pod linkiem https://pydicom.github.io/pydicom/stable/#. Oczywiście można też skorzystać z wersji dostępnej na Github: https://github.com/pydicom/pydicom
Na początek musimy tagi pobrać z obrazu, stąd musimy mieć jakieś dane z rozszerzeniem .dcm. Można je pobrać z Kaggle, ale wymagane jest założenie konta. Na dłuższą metę konto się przydaje, bo Kaggle ma do zaoferowania mnóstwo datasetów, na którym możesz się wiele nauczyć. Później też możesz wchodzić do konkursu.
Odbiegłam trochę od tematu, wracamy do naszego datasetu. Dostępny jest on pod adresem https://www.kaggle.com/datasets/kmader/siim-medical-images/data. Po pobraniu i wypakowaniu danych wejdź do folderu dicom_dir i tam będą interesujące nas pliki.
Następnie musimy zapisać gdzieś/zapamiętać ścieżkę podczas tworzenia programu. Będą nam potrzebne dwie biblioteki: pydicom oraz matplotlib.pyplot.
Oczywiście później trzeba dodać funkcję wyświetlania i voila.
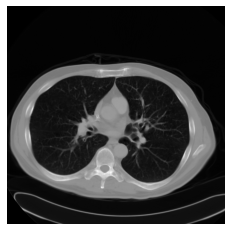
W efekcie otrzymujemy i obraz:
oraz tagi
Z czego PixelData i przede wszystkim PixelArray przydaje nam się do rekonstrukcji obrazu z projekcji, ale o tym w innym wpisie.


Komentarze
Prześlij komentarz